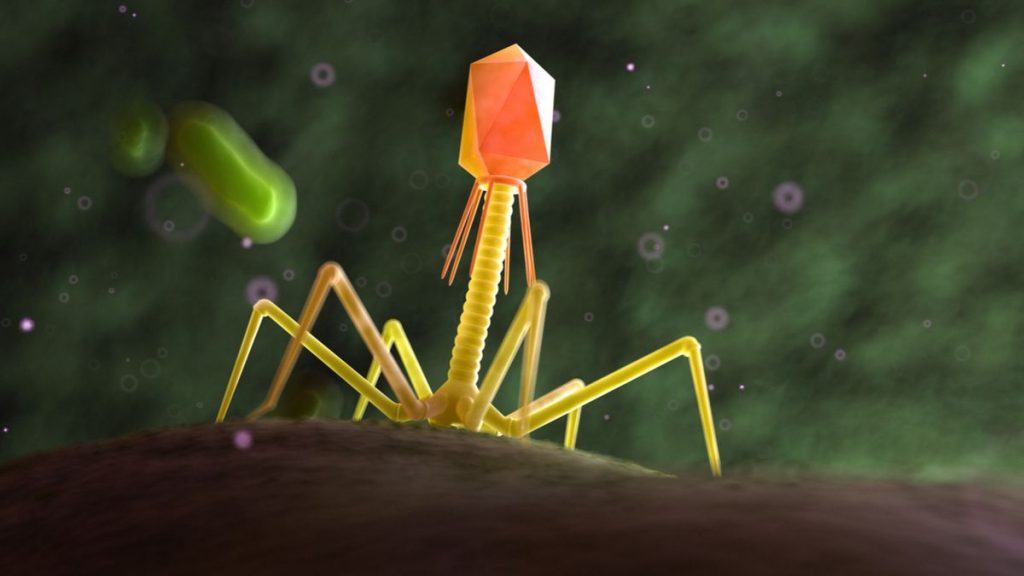
Une série de recherches a récemment montré que certains virus chasseurs de bactéries utilisaient un alphabet génétique que l’on ne retrouve nulle part ailleurs sur notre planète. Explications.
Un probable mécanisme d’autoprotection
Les virus s’attaquant aux bactéries (appelés bactériophages) et leurs proies sont en guerre depuis des lustres, chaque camp développant des tactiques avancées pour infecter ou détruire l’autre. Finalement, certains bactériophages ont porté cette course aux armements à un niveau supérieur en modifiant la façon dont ils codent leur ADN.
L’ADN génomique est composé de quatre nucléotides standards. Ces nucléobases forment l’alphabet génétique ATCG, conservé dans tous les domaines de la vie. Cependant, en 1977, des chercheurs ont découvert que le cyanophage S-2L, virus à ADN, avait remplacé toutes les occurences de « A » par de la 2-aminoadénine (Z) dans l’ensemble de son génome, formant ainsi l’alphabet génétique ZTCG.
La base « Z » formant une triple liaison avec la base « T » opposée (soit une de plus que pour la connexion A:T normale) dans les « barreaux » de connexion de la double hélice d’ADN, les chercheurs avaient à l’époque conclu qu’il s’agissait d’un mécanisme d’autoprotection, rendant le génome viral plus résistant et plus difficile à séparer pour les bactéries à l’aide de substances chimiques appelées nucléases.

Bien que les scientifiques aient été fascinés par cette découverte, aucun autre bactériophage à génome Z n’avait été identifié au cours des décennies suivantes. Une série de recherches publiées dans la revue Science a récemment permis de confirmer qu’il ne s’agissait pas d’un cas unique, tout en caractérisant le fonctionnement et l’assemblage du génome Z.
De nombreux bactériophages concernés
Deux équipes respectivement dirigées par Yan Zhou, chercheur à l’université de Tianjin, et Dona Sleiman, microbiologiste à l’Institut Pasteur, ont identifié deux protéines majeures, appelées PurZ et PurB, constituant la base « Z ». Tandis qu’un troisième groupe, dirigé par Valérie Pezo, de l’université Paris-Saclay, a corroboré ces résultats et procédé à l’analyse de l’enzyme DpoZ, responsable de l’assemblage de l’ensemble du génome Z.
« Les scientifiques rêvent depuis longtemps d’augmenter la diversité des bases. Notre travail montre que la nature a déjà trouvé un moyen d’y parvenir », a souligné Zhou.
La recherche de séquences génétiques associées à ces protéines et enzymes dans différentes bases de données a ensuite montré qu’une grande variété de bactériophages possédaient des gènes similaires.
« Notre travail pourrait éveiller un intérêt pour la recherche interdisciplinaire sur les origines de la vie et l’astrobiologie »
Pour les scientifiques, la prochaine étape consistera à explorer la compatibilité du génome Z avec une machinerie cellulaire ordinaire telle que la nôtre ainsi que ses éventuelles applications.
« La base Z a été identifiée sans ambiguïté dans une météorite carbonée et proposée comme une nucléobase qui aurait pu être disponible pour l’origine de la vie », a ajouté Zhou. « Une telle provenance suggère que notre travail pourrait éveiller un intérêt pour la recherche interdisciplinaire sur les origines de la vie et l’astrobiologie. »
Par Yann Contegat, le
Source: Science Alert
Étiquettes: adn, virus, bactérie, genome, z, bactériophage
Catégories: Sciences, Actualités

Les purines modifiées ont beaucoup à nous apprendre sur les mécanismes de résistance au stress !